Boa tarde pessoal.
Recebi o seguinte erro ao executar o código no RStudio:
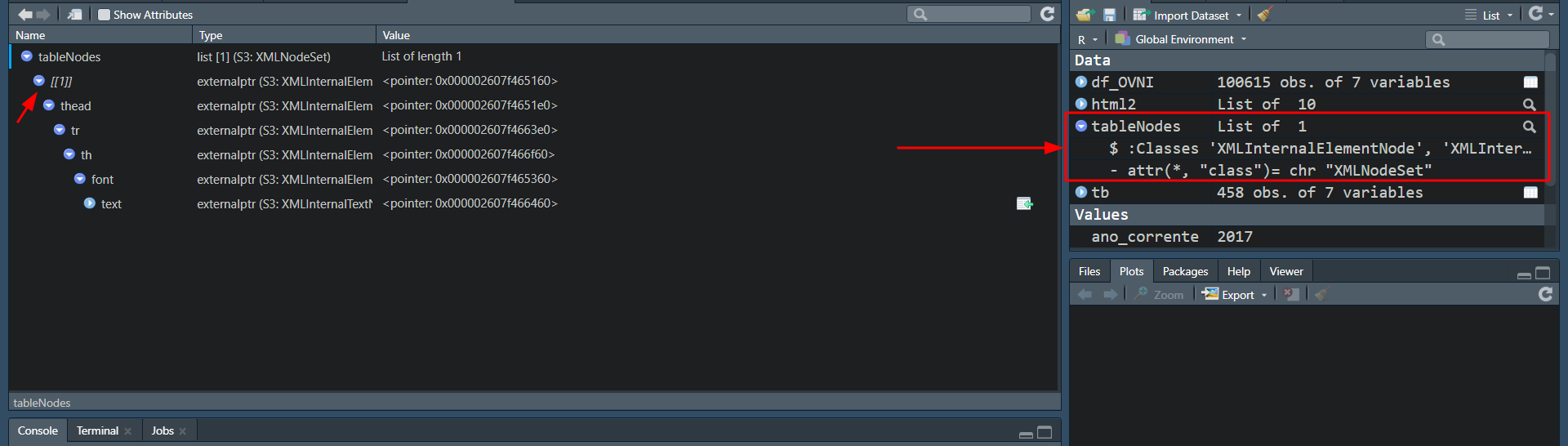
Error in tableNodes[[1]] : subscript out of bounds
O código que estou executando é este:
while (ano_mes_corrente <= 201709) {
site <- paste("http://www.nuforc.org/webreports/ndxe", as.character(ano_mes_corrente), ".html")
site <- gsub (" ", "", site)
html2 <- GET(site)
parsed <- suppressMessages(htmlParse(html2, asText=TRUE))
tableNodes <- getNodeSet(parsed, "//table")
tb <- readHTMLTable(tableNodes[[1]])
df_OVNI <- rbind(df_OVNI,tb)
if (mes_corrente == 12)
{
mes_corrente <- 1
ano_corrente <- ano_corrente + 1
ano_mes_corrente <- (ano_corrente * 100) + mes_corrente
}
else
{
mes_corrente <- mes_corrente + 1
ano_mes_corrente <- ano_mes_corrente + 1
}
print(ano_mes_corrente)
}
Desde já agradeço.
Flávia